library(canaper)
library(tidyverse)
# 再現のためにシードを設定する
set.seed(12345)
# 1. ランダム化比解析を行う
acacia_rand_res <- cpr_rand_test(
acacia$comm, acacia$phy,
null_model = "curveball",
n_reps = 99, n_iterations = 10000,
tbl_out = TRUE
)
# 2. 固有性を分類化する
acacia_canape <- cpr_classify_endem(acacia_rand_res)
canaperへの紹介
(Read this blogpost in English)
canaper v1.0.0がCRANに登録されました!今までいくつかのRパッケージを書いてGitHubで公開したことがありますが、自分のパッケージがCRANに登録されるのが初めてです。
そもそも、canaperって一体何だろう?
パッケージのDESCRIPTIONにはこのような文章があります:
canaperprovides functions to analyze the spatial distribution of biodiversity, in particular categorical analysis of neo- and paleo-endemism (CANAPE) as described in Mishler et al (2014) doi:10.1038/ncomms5473.canaperconducts statistical tests to determine the types of endemism that occur in a study area while accounting for the evolutionary relationships of species.
つまり、canaperは生物多様性の地理的分布を解析する関数を提供します。特に、生物の進化的な関係を考慮しながら、ある地域における固有性の種類を統計的に検証する、categorical analysis of neo- and paleo-endemism(CANAPE)というMishler等(2014)doi:10.1038/ncomms5473 が開発した解析を行います。
もしも上の話を読んで「面白い!」と思ったら、是非続きを読んでください。
系統的固有性とCANAPE
生物多様性は種数、つまり、ある地域における種の数で測られることがよくあります。それと同じように、固有性はある地域にしか生息しない種の数で測ることがよくあります。しかし、このような、種名だけを使うアプローチは種の進化的な歴史を考慮しません。最近、分子系統樹の増加によって、種の進化的な歴史を考慮した生物多様性を測定する方法がいくつか開発されました。その一つは系統的固有性(Phylogenetic endemism, PE; Rosauer ほか 2009)です。PEは種ではなく、系統樹の枝によって固有性を測る方法です。
PEを使うことによって、生物多様性を生み出す進化的なプロセスを垣間見ることができる。例えば、PEの高い、短い枝が密集している地域は最近の種分化(放散)によってできた可能性があり、このような地域を「neo-endemic」と呼びます。一方で、PEの高い、長い枝が密集している地域はかつて広く分布していた系統が多く絶滅したことによってできた可能性が高く、このような地域を「paleo-endemic」と呼びます。このような地域の区別をするために、Mishler ほか (2014) がCANAPEという方法を開発しました。
canaperの目的はRでCANAPEを行うことです。
実例:オーストラリアのアカシア

canaperには元々のCANAPEの論文で解析されたデータセットが備えられています。オーストラリア産のアカシア1 のデータです。系統樹とコミュニティマトリックス(群集⾏列)からなっています。このデータセットを用いて簡単なデモを行います2。
ここで詳細には入りませんが、この例についてもっと知りたければ、canaperのウェブサイトをご覧ください。
CANAPE解析の全部を二つのコマンドだけでできます:cpr_rand_test()と cpr_classify_endem()。
では、アウトプットをみてみましょう。
cpr_rand_testがたくさん(全部で54)の列を返します。コミュニティの各地点について、様々な(PEを含めた)指標です。
acacia_rand_res# A tibble: 3,037 × 55
site pd_obs pd_rand_mean pd_rand_sd pd_obs_z pd_obs_c_upper pd_obs_c_lower
<chr> <dbl> <dbl> <dbl> <dbl> <dbl> <dbl>
1 -1025… 0.0145 0.0227 0.00506 -1.62 1 98
2 -1025… 0.0382 0.0497 0.00745 -1.54 8 91
3 -1025… 0.0378 0.0369 0.00635 0.138 55 44
4 -1025… 0.0570 0.0613 0.00829 -0.517 33 66
5 -1025… 0.0409 0.0419 0.00614 -0.172 41 58
6 -1025… 0.00998 0.0101 0.00193 -0.0429 49 48
7 -1025… 0.0187 0.0225 0.00393 -0.958 19 80
8 -1025… 0.0434 0.0536 0.00902 -1.14 15 84
9 -1025… 0.0111 0.0101 0.00197 0.495 81 18
10 -1025… 0.0903 0.0876 0.0112 0.240 61 38
# ℹ 3,027 more rows
# ℹ 48 more variables: pd_obs_q <dbl>, pd_obs_p_upper <dbl>,
# pd_obs_p_lower <dbl>, pd_alt_obs <dbl>, pd_alt_rand_mean <dbl>,
# pd_alt_rand_sd <dbl>, pd_alt_obs_z <dbl>, pd_alt_obs_c_upper <dbl>,
# pd_alt_obs_c_lower <dbl>, pd_alt_obs_q <dbl>, pd_alt_obs_p_upper <dbl>,
# pd_alt_obs_p_lower <dbl>, rpd_obs <dbl>, rpd_rand_mean <dbl>,
# rpd_rand_sd <dbl>, rpd_obs_z <dbl>, rpd_obs_c_upper <dbl>, …cpr_classify_endem()がもう一つの列をデータに付けます。新しい列は固有性の種類です。それぞれの種類が何回観察されたのか、数えてみましょう:
count(acacia_canape, endem_type)# A tibble: 5 × 2
endem_type n
<chr> <int>
1 mixed 176
2 neo 5
3 not significant 2827
4 paleo 12
5 super 17そして、今回計算した固有性の種類を地図にするとこうなります:
コード
# まず、図を作るためにデータをちょっといじる
# (経緯度の列を加える)
acacia_canape <- acacia_canape |>
separate(site, c("long", "lat"), sep = ":") |>
mutate(across(c(long, lat), parse_number))
# 図のテーマをいじる
theme_update(
panel.background = element_rect(fill = "white", color = "white"),
panel.grid.major = element_line(color = "grey60"),
panel.grid.minor = element_blank()
)
ggplot(acacia_canape, aes(x = long, y = lat, fill = endem_type)) +
geom_tile() +
# cpr_endem_cols_4 はcanaperに入っているカラーユニバーサルデザインのパレット
scale_fill_manual(values = cpr_endem_cols_4) +
coord_fixed() +
guides(
fill = guide_legend(title.position = "top", label.position = "bottom")
) +
theme(legend.position = "bottom", legend.title = element_blank())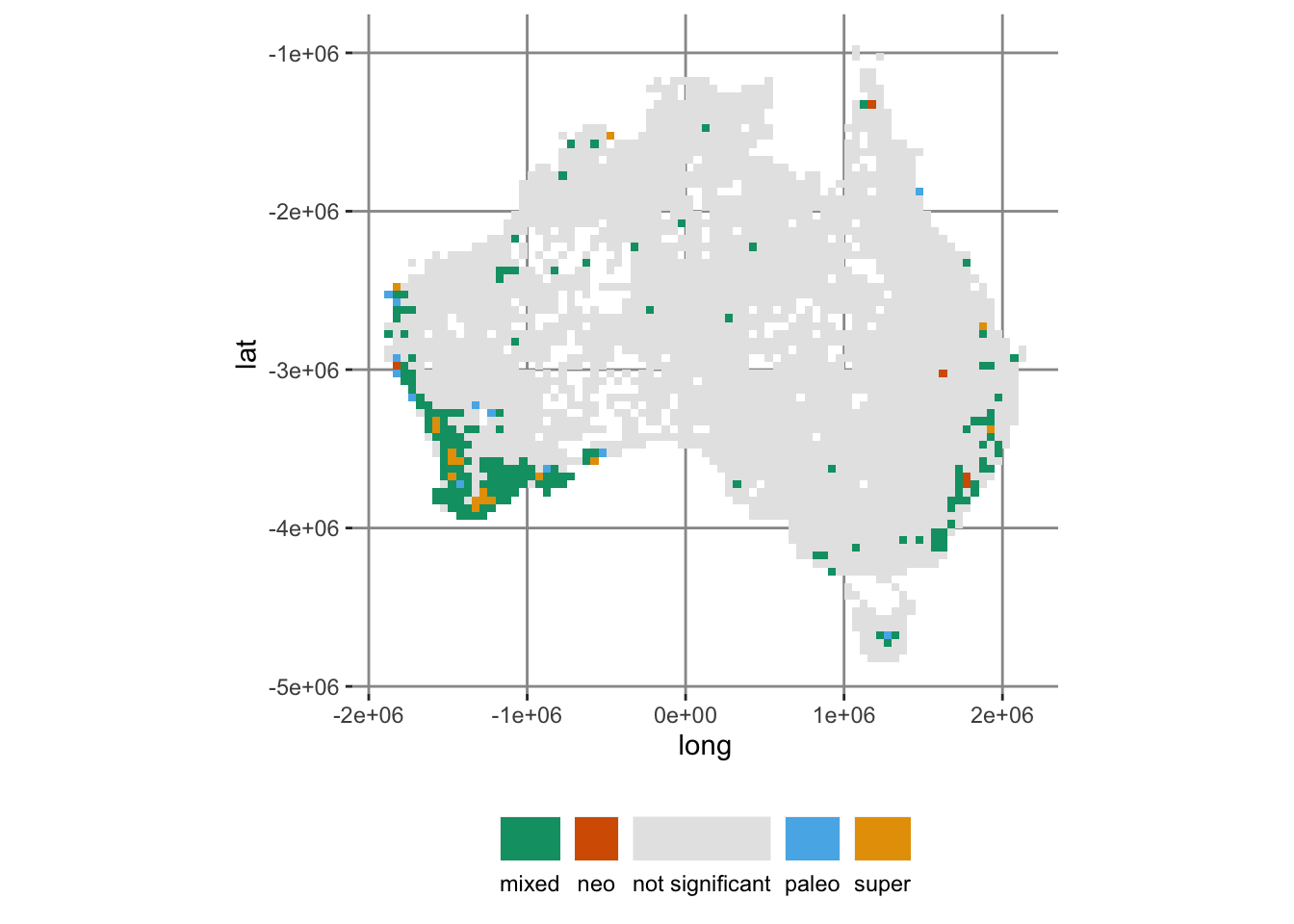
rOpenSci
もう一つ今回で初めてだったのがROpenSciにパッケージを投稿することでした。rOpenSciはRで書かれた研究用のソフトを支援する団体です。もし自分の研究用のパッケージを公開しようと考えているなら、とてもおすすめです。
というのは、まずはRパケージの書き方について非常に丁寧な説明書を提供しているからです。また、パッケージの自動的なチェックを行うパッケージも。これを使うだけでも自分のコードの腕がかなり上がりました。
次に、rOpenSciに投稿されたパッケージは徹底的なコードレビュー(査読)を受けることになっています。こうすることによって、自分だけではなかなか気づかなかったことを教えていただき、さらにコードの改善につながりました3。
しかし、何と言っても、やはりrOpenSciのコミュニティが素晴らしいです。とてもアクティブで広く開かれたコミュニティです。活動としては、Community Call(誰でも参加できるビデオコール)、バーチャルコーワークスペース、やSlackのチャットチャンネルがあります。
ご興味のある方は是非試してくださいね!そして、rOpenSciに感謝します!
参考情報
canaperについてもっと知りたい方はGitHubのサイト、パッケージのサイト、およびプレプリントをご覧下さい。
参考文献
再現性
脚注
アカシアはオーストラリアの被子植物の中で最も種が多い属です。1000種近くあります。↩︎
なお、本デモに使う解析設定は都合上使っているだけで、本格的な解析にはおすすめしません。↩︎
査読者のKlaus SchliepとLuis Osorio、そして編集者のToby Hockingに感謝しています!↩︎
ライセンス
引用
@online{2022,
author = {},
title = {canaperがCRANに登録されました},
date = {2022-10-07},
url = {https://www.joelnitta.com/posts/canaper/},
langid = {ja}
}